U-Net (Convolutional Networks for Biomedical Image Segmentation) 리뷰

Abstract(요약)

- 딥러닝 네트워크를 성공적으로 훈련하기 위해서 training set 필요.
- training set 샘플의 효과적 학습을 위한 data augmentation(데이터 증강) 제시
- 모델링은 특징 추출을 위한 "contracting path" 와 정확한 localization을 위한 "expanding path"로 이루어짐
- contracting path-expanding path의 네트워크는 적은 수의 이미지로도 end-to-end학습 가능, 기존 convolutional network보다 성능 더 효과적
- 2015 ISBI cell tracking대회에서 U-Net 성능 입증
1 Introduction
- convolutional network task : single class label 도출
- Biomedical task : 이미지 class 도출 + localization(각 픽셀에 클래스 label 할당되어야 함)
- localization문제 : 논문에서 언급한 방식은 pixel과 pixel 주변의 영역정보를 받고 객체 식별 및 판단하는 방식. 이런 방식은 학습데이터의 단위가 이미지단위가 아닌 이미지 patch가 단위로 설정 -> 더 많은 데이터셋 구성
- patch 단점 :
- patch별로 연산하게 되며, 연산속도 느려짐 + 중복 영역 patch의 결과로 중복 예측결과 발생
- patch가 클수록 max-pooling작업 필요, localization 정확도 낮아지는 문제 발생, patch가 작을수록 특징 제대로 추출할 수 없는 trade-off 발생
- trade-off : 하나의 선택을 위해 다른 선택 포기해야하는 상황
- patch단점 해결하기 위해 개선한 U-Net 제시
- fully convolutional network구조로 기존보다 탄력적인 구조
- patch 단점 :
U-Net의 모델 : 적은 데이터셋에 학습시켜도 좋은 성능 도출 가능
U-Net의 구조 : 특징 추출하여 점차 해상도가 줄어드는 feature map은 up-sampling, 이전에 얻은 feature map과 이어주는 과정
- up-sampling : 이미지를 확대하여 해상도를 높이는 과정, 예를 들어, 64x64 픽셀의 이미지를 128x128 픽셀로 확대
- Channel(채널): 이미지 데이터에서 색상 정보를 담는 단위

- U-net의 특징
1) up-sampling과정에서 channel의 수가 더 많기 때문에 higher resolution layer에 context information을 전파
- up-sampling을 통해 해상도가 높아진 이미지에서 채널 수가 많으면, 더 많은 세부 정보와 컨텍스트를 포함할 수 있어서 원본 이미지의 정보를 더 잘 유지할 수 있음
- higher resolution layer : 해상도가 더 높아진 레이어
2) contracting path(점차 해당도가 줄어드는 경로) 와 expansive path(점차 해상도가 증가하는 경로) 의 layer 개수가 거의 대칭이 되기 때문에 U자 형태를 띔
3) Fully connected layer를 사용하지 않았기 때문에, patch에서 얻은 정보만을 가지고 해당 patch에서 classification을 수행
- FC layer를 없애고 마지막에 Fully convolution layer로 마무리 짓는 이유 : model architecture 참조
4) Data augmentation을 사용
: 이러한 Data augmentation(데이터 증강)은 Biomedical segmentation(식별 및 분할)에서 중요한데, deformation(변형)이 조직(근육조직 등)의 공통적 variation으로 가장 많이 쓰이는 방식이고 사실적 deformation은 효과적으로 시뮬레이션 될 수 있기 때문이라고 한다.
5) 세포의 경우, 세포가 여러개 붙어있기에 구분하기 힘들다는 문제. 이를 U-net은 거리를 이용한 가중치를 통해 만든 loss function으로 극복
6) U-net은 Semantic Segmentation에서 다른 모델과 비교했을 때 좋은 성능 입증
2 Network Architecture


Blue tile과 Yellow tile이 서로 Overlap되어 있음을 확인할 수 있으며, Blue area에 기반하여 Yellow area의 Segmentation을 prediction 했음을 확인 할 수 있다. 그 이후, missing data는 mirroring을 통하여 extrapolation(외삽)
여기서, extrapolation은 한국어로 "외삽한다" 라고 표현하는데, 외삽한다 라는 의미는 다른 변수와의 관계에 기초하여 변수 값을 추정한다는 것
Pooling을 할 때, 0으로 이루어진 값으로 인해 값이 사라지게 되는데 이 사라진 값을 추정하겠다는 의미
Contracting path(수축 경로)
- 수축 경로는 2개의 3X3 conv를 반복적으로 적용, 각각의 활성화 함수(Relu-recifited linear unit)와 다운샘플링(stride=2, 2X2 max-pooling) 진행
- 2x2 max-pooling을 위해 x와 y의 input tile size 짝수로 설정
- 다운샘플링 단계마다 피쳐 채널 수를 두 배로 늘림
- 주로 컨볼루션 레이어와 풀링 레이어를 사용하여 이미지를 압축
- padding없이 연산 진행,
Expansive path(확장 경로)
- 확장 경로에서 각 단계는 feature map의 업샘플링과 특징 채널 수를 절반으로 줄이는 2X2 conv 구성
- up-sampling은 Interpolation와 Deconvolution 이용
- Interpolation : 확장 후 중간값 매꾸기
- Deconvolution : 파라미터를 이용해 확장-겹치는 영역 더하기
- up-sampling은 Interpolation와 Deconvolution 이용
- localization(식별)을 위해, 위치정보가 들어있는 contracting path의 feature map을 crop(잘라서)하여 expansive path에 붙이기
- 맨 마지막 단계에 1x1 convolution을 사용하여 각 64개 컴포넌트 특징 벡터를 원하는 수(논문에서는 2개: foreground, background)의 클래스로 매핑

Q. 그렇다면 U-net은 왜 Fully Convolution layer로 dense layer(연결 레이어)를 사용하지 않을까?
Segmentation task(분할 식별작업)는 세분화된 픽셀별로 클래스를 정하기에 위치정보가 굉장히 중요. 하지만 이러한 위치정보가 dense layer에 들어가면서 사라짐. Segmentation task는 세분화된 픽셀단위 예측이 필요
- dense layer에 들어가면 위치정보가 사라지는 이유
- flatting(평탄화) : 이미지를 1차원 벡터로 변환, 이 과정에서 각 픽셀의 위치 정보가 손실
- 위치 비의존적 연산 : Dense Layer는 입력 벡터의 각 요소를 동일하게 취급, 이미지의 공간적 구조와 상관없이 연산이 이루어짐
-> 따라서, Dense layer가 아닌, conv layer(Fully Convolution layer)를 들어가게함으로써 해결
- 컨볼루션 레이어는 이미지를 처리하면서 공간적 구조를 유지한 채, 연산 진행
- 컨볼루션 레이어는 국소적 연산을 수행함으로, dense layer보다 연산 효율 높음
U-Net 구조 요약
- 수축 경로: 컨볼루션 -> ReLU -> 컨볼루션 -> ReLU -> 맥스 풀링
- 확장 경로: 업샘플링 -> 컨캣 -> 컨볼루션 -> ReLU -> 컨볼루션 -> ReLU
3 Training
- GPU를 최대한 활용하기 위해, 이미지 파일을 여러개의 batch로 나눔
- 학습용 데이터 셋의 단위는 이미지의 '일부'
- 필요한 이미지 개수는 줄이면서 데이터 셋 내 입력 파일의 개수는 늘림
Loss function
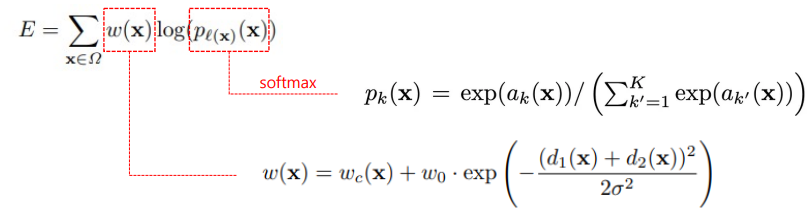
- a_k(x) : 픽셀 x가 class k일 값(픽셀 별 모델의 Output)
- p_k(x) : 픽셀 x가 class k일 확률(0~1)
- l(x) : 픽셀 x의 실제 label
- w0 : 논문의 Weight hyper-parameter, 논문에서 10으로 설정
- σ : 논문의 Weight hyper-parameter, 논문에서 5로 설정
- d1(x) : 픽셀 x의 위치로부터 가장 가까운 경계와 거리
- d2(x) : 픽셀 x의 위치로부터 두 번째로 가까운 경계와 거리
- U-net에서 loss function은 cross entropy를 사용
- 그림처럼 세포간의 경계를 명확하게 하기 위한 추가적인 가중치 w(weight map loss)가 포함
- 거리가 경계와 가까우면 loss값이 커짐
- loss = weight map(w) X log(픽셀에서 얻은 클래스별 예측값을 softmax함)
3.1 Data Augmentation
- Data augmentatoin(데이터 증강)은 주로 데이터 셋이 많지 않을 때 사용
현미경 등으로 촬영하는 이미지들은 색깔이 다양하지 않고, 회색빛으로 이루어져있기에 객체 간에 구별이 선명하지 않음. 따라서 Data augmenatation을 이용하여 풍부한 데이터 셋을 만듬
=> 이를 통해 invariance하고 rubustness한 성질을 갖는 모델로 학습
U-net은 Data augmentation을 위해 shift, rotation random-elastic deformation을 수행
특히 논문에서 "random-elastic deformation(탄성 변형)" 을 작은 데이터 셋을 가지고 segmentation network를 학습시킬 때 key concept으로 여긴다고 하였기에 주목할 필요가 있음
- Elastic Deformation(탄성 변형) 사용을 추천하는 경우 :
연속체에서 어떠한 힘이나 시간의 흐름으로 인해 변화가 발생하는 경우.
이 힘이 제거된 후 변형이 원래처럼 돌아오게 되면 이 변형을 '탄성'이라고 한다. 이렇게 탄성이 있는 경우는 같은 물체라고 해도 촬영 방법이나 각도 등에 의해서 다른 결과를 가져올 수 있으므로 이럴 때 사용하면 좋다고함

이렇게 생성된 이미지는 원본 이미지와 형태가 유사하지만 약간의 찌그러짐이나 늘어남을 가짐
이러한 변형된 이미지들을 훈련 데이터셋에 추가함으로써 모델은 이러한 변형에 대해 더 강인한 특성을 학습할 수 있고, 이는 모델의 일반화 성능을 향상시키며 데이터의 다양성을 높여 새로운 입력에 대한 예측 능력을 향상시킬 수 있음
4 Experiments
Table1. EM segmentation challenge
전자현미경으로 관찰되는 뉴런 구조에서 cell segmentation task를 수행
데이터셋은 전자 현미경으로 찍은 512 x 512 해상도의 이미지 30장으로 이루어져 있고, 이미지의 각 부분에 세포는 흰색, 세포막은 검은색으로 칠해짐
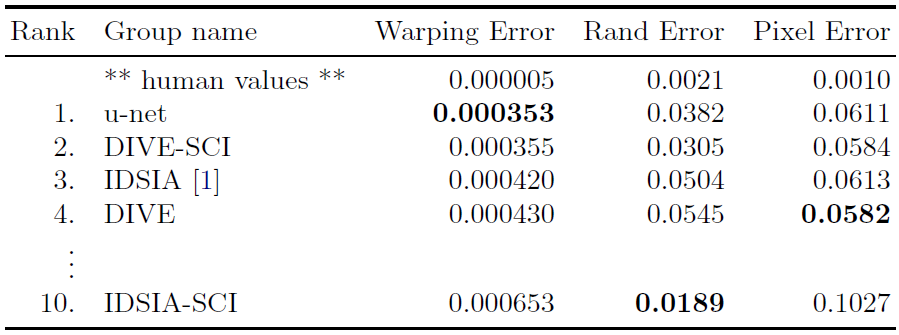
- Pixel Error : prediction과 ground truth에 pixel마다 할당된 class들끼리 불일치하는 pixel들의 전체 pixel 기준 비율
- Warping Error & Rand Error : digital topology error 관련
U-net을 포함한 10개의 모델을 가지고 성능을 평가했고, 결과
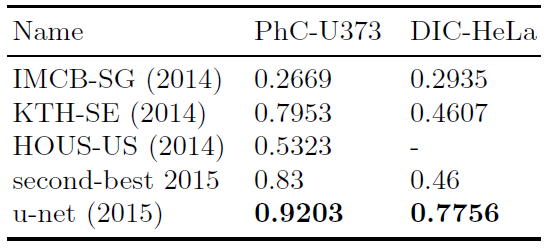
Table2. Segmentation results (IOU) on the ISBI cell tracking challenge 2015.
광학 현미경(light microscopic)에서 얻은 이미지로도 cell segmentation task를 수행
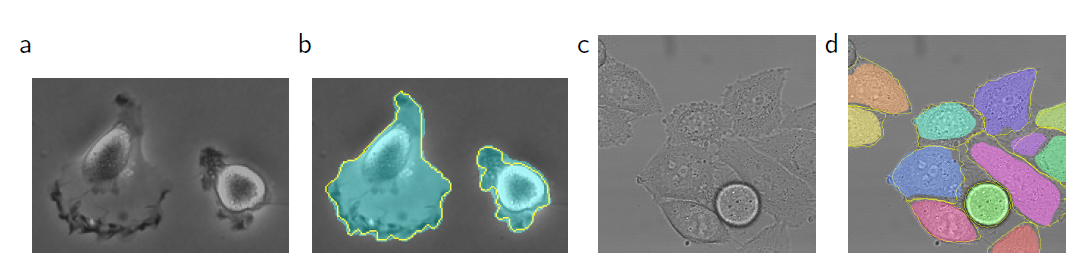
a와 c가 입력 이미지이며, b와 d가 ground truth segmentation map
a와 c같은 이미지를 통해 세포를 얼마나 잘 구별할 수 있는지 시험
성능은 IOU로 측정, 결과는 아래
5 Conclusion
- U-net은 U자형 아이디어 모델링을 통해 biomedical segmentation(분할식별) 분야에서 이전보다 좋은 성능 입증
- Data augmentation에서 사용한 Elastic Deformation(탄성변형) 기법 덕분에 적은 사이즈의 데이터셋만 요구, 합리적 학습시간 사용